-

姓 名:李富义
职 称:教 授
办公室:det365官方网站登录414
邮 箱:fuyi.li@nwafu.edu.cn
最新消息 · 最新接收论文: Li F*, Bi Y, Guo X, Tan X, Wang C, Pan S. "Advancing mRNA subcellular localization prediction with graph neural network and RNA structure". Bioinformatics, 40(8), btae504.
· 最新接收论文(博士生一作): Bi Y, Li F*, Wang C, Pan T, Davidovich C, Webb Gl*, Song J*. "Advancing microRNA Target Site Prediction with Transformer and Base-Pairing Patterns". Nucleic Acids Research. qkae782.
· 最新接收论文(研究生一作): Liu T, Jia C*, Bi Y, Guo X, Zou Q*, Li F*, “scDFN: enhancing single-cell RNA-seg clustering with deep fusion networks". Briefings in Bioinformatics, 25(6), bbae486.
· 最新接收论文(研究生一作): Zhang Z, Liu Y, Xiao M, Wang K, Huang Y, Bian J, Yang R*, Li F*, "Graph Contrastive Learning as a Versatile Foundation for Advanced scRNA-seg Data Analysis". Briefings in Bioinformatics. in press.
· 最新接收论文(研究生一作): Jia R, He Z, Wang C, Guo X, Li F*, “MetalPrognosis: a Biological Language Model-based Approach for Disease-Associated Mutations in Metal Binding Site Prediction". lEEE/ACM Transactions on Computational Biology and Bioinformatics. in press.
· 最新接收论文(本科生一作): Zhao Z, Shi G, Wu X, Ren R, Gao X*, Li F*. “DIG-Mol: A Contrastive Dual-Interaction Graph Neural Network for Molecular Property Prediction". lEEE Journal of Biomedical and Health Informatics. in press.
· 最新接收论文(研究生冉子旭一作):Ran Z, Wang C, Sun H, Pan S, Li F*. “Characterizing secretion system effector proteins with structure-aware graph neural networks and pre-trained language models”. IEEE Journal of Biomedical and Health Informatics. in press. (IF=7.7, 中科院大类一区, Top期刊)
· 李富义教授入选美国斯坦福大学和爱思唯尔数据库(Elsevier Data Repository)联合发布的2023年全球前2%顶尖科学家榜单(Stanford University Top 2 Scientists List),数据网站:https://elsevier.digitalcommonsdata.com/datasets/btchxktzyw/4;排名查询:https://jokergoooo.shinyapps.io/top2pct_scientists/_w_293d46a8/#
· 最新接收论文(研究生王聪一作):Wang C, He Z, Jia R, Pan S, Coin LJM, Song J, Li F*. “PLANNER: a multi-scale deep language model for the origins of replication site prediction”. IEEE Journal of Biomedical and Health Informatics. in press. (IF=7.7, 中科院大类一区, Top期刊)
· 最新接收论文:Li F*, Wang C, Guo X, Akutsu T, Webb GI, Coin LJM, Kurgan L*, Song J*. “ProsperousPlus: An integrated platform for protease-specific substrate cleavage prediction and machine learning model construction of more than 100 proteases”. Briefings in Bioinformatics. 2023, 24(3), bbad170. (IF=13.994, 中科院大类一区, Top期刊, CCF B类, 西农G2期刊)
· 最新成果“ProsperousPlus: An integrated platform for protease-specific substrate cleavage prediction and machine learning model construction of more than 100 proteases”获评生物信息学顶会ISMB/ECCB的Royal Society of Chemistry Best Poster Prize (https://www.iscb.org/ismbeccb2023-general-info/awards-winners).
· 最新接收论文:Chen R†, Li F*†, Guo X, Bi Y, Li C, Pan S, Coin LJM*, Song J*. “ATTIC is an integrated approach for predicting A-to-I RNA editing sites in three species”. Briefings in Bioinformatics. 2023, 24(3), bbad170. (IF=13.994, 中科院大类一区, Top期刊, CCF B类, 西农G2期刊),该研究成果被det365官方网站登录官网报道:https://news.nwsuaf.edu.cn/xscg/d65de30fa07e4456beb22c060229437b.htm
· 团队开发的世界上第一款针对结合分枝杆菌PE/PPE蛋白家族的专业在线数据库正式上线:http://merits.unimelb-biotools.cloud.edu.au/,欢迎大家访问使用
· 最新接收论文:Li F*, Guo X, Bi Y, Jia R, Pitt ME, Pan S, Li S, Gasser RB, Coin LJM*, Song J*. “Digerati – a multipath parallel hybrid deep learning framework for the identification of mycobacterial PE/PPE proteins”. Computers in Biology and Medicine. 2023, 163, 107155. (IF=6.698, 中科院大类二区, 小类一区)
· 最新接收论文:Zhu Y†, Li F*†, Guo X, Wang X, Coin LJM, Webb GI, Song J*, Jia C*. “TIMER is a Siamese neural network-based framework for identifying both general and species-specific bacterial promoters”. Briefings in Bioinformatics. 2023, in press. (IF=13.994, 中科院大类一区, Top期刊, CCF B类, 西农G2期刊)
· 论文“ProsperousPlus: An integrated platform for protease-specific substrate cleavage prediction and machine learning model construction of more than 100 proteases”被ISMB/ECCB国际会议 (CCF B类会议) 接受并邀请做Oral Presentation
· 恭喜硕士生Ruyi Chen (墨尔本大学生物信息学) 荣获澳大利亚昆士兰大学博士全额奖学金攻读博士学位!
· 受邀作为Keynote Speaker在第二届电子信息工程与计算机技术国际会议(The 2nd International Conference on Electronic Information Engineering and Computer Technology)上做大会报告:“Developing data-driven machine learning approaches to address bioinformatics classification problems”
· 最新接收论文:Bi Y, Li F*, Guo X, Wang Z, Pan T, Guo Y, Webb GI, Yao J, Jia C*, Song J*. “Clarion is a multi-label problem transformation method for identifying mRNA subcellular localisations”. Briefings in Bioinformatics. 2022, in press. (IF=13.994, 中科院大类一区, Top期刊, CCF B类, 西农G2期刊)
· 受邀担任BMC Bioinformatics (生物信息学老牌杂志,CCF C类) 编委
· 受邀担任计算生物学和生物医学国际学术会议 (CBBS 2023)程序委员会委员http://www.iccbbs.org/CommitteeCn.aspx
· 最新接收论文:Liu Q, Fang H, Wang M, Li S, Coin LJM, Li F*, Song J*. “DeepGenGrep: a general deep learning-based predictor for multiple genomic signals and regions”. Bioinformatics. 2022, 38(17):4053–4061. (IF=6.937, Top期刊, 中科院大类二区, CCF B类),论文被人工智能和生物信息学公众号AI in Graph (https://mp.weixin.qq.com/s/vrEU7kjmWAWg_DelKK_Kog) 和 DrugAI (https://mp.weixin.qq.com/s/X-6eON66dtrwGRUhznbbQQ) 公开报道
基本信息 李富义,男,博士,教授,博士生导师,澳大利亚墨尔本大学医学、牙医与健康科学学院(Faculty of Medicine, Dentistry and Health Sciences, University of Melbourne)荣誉研究员(Honorary Fellow),中国计算机学会(CCF)会员,澳大利亚生物信息学和计算生物学学会(ABACBS)会员,澳大利亚生物化学和分子生物学学会(ASBMB)会员。2020年5月于澳大利亚蒙纳士大学(Monash University)生物医学发现研究所获得博士学位,博士期间师从国际生物信息学专家 Jiangning Song教授和澳大利亚科学院院士、蒙纳士大学抗菌素耐药性研究中心主任Trevor Lithgow教授,毕业后加入澳大利亚墨尔本大学(The University of Melbourne)Peter Doherty感染与免疫研究所担任生物信息学研究员(Level B)。2022年6月起任det365官方网站登录长聘教授。
主要研究方向为机器学习、数据挖掘和生物信息学等。近年来在Nature Communications, Nucleic Acid Research, Bioinformatics, Briefings in Bioinformatics, Genomics Proteomics & Bioinformatics和Molecular Microbiology 等国际知名期刊上发表论文63篇(中科院大类一区30篇),其中有12篇文章被Clarivate评为高被引文章,2篇为热点文章,Google Scholar总引用次数达到3213次(H-index 42, i10-index 27, 2023年5月)。个人多篇一作文章受到澳大利亚科学院院士Trevor Lithgow教授,澳大利亚科学院院士Jian Li教授,Monash数据未来研究所主任Geoff Webb教授(IEEE fellow),京都大学生物信息学中心Tatsuya Akutsu教授等相关领域权威专家的认可与肯定。近年来,研发了二十多款生物信息学数据库、软件和分析平台,这些工具在过去五年内得到了80多个国家和地区的国际用户超过230,000次的使用,这些生物信息学软件已经被发表在包括Cell, PNAS, Nature Chemical Biology和Nucleic Acids Research等国际高水平期刊的研究工作引用2000余次,产生了较为广泛的影响力。
团队招生信息 课题组诚招具有责任心、学习积极主动和具有较强的自律性的博士后加盟,欢迎硕士和博士生推免报考!有意加盟团队的优秀学者学子,请与我邮件联系。
博士生招生专业方向:农业工程
硕士生招生专业方向:计算机科学与技术(学硕)
计算机技术、人工智能 (专硕)
团队和澳大利亚Monash大学、墨尔本大学、格里菲斯大学、迪肯大学,日本京都大学等国际一流高校保持长期合作开展科学研究,学术资源丰富,能为学生的成才和发展提供强有力的支持和帮助。
团队将为你提供:
· 前往国内外知名大学进行学术交流的资助和机会。团队与很多国际一流的研究团队保持长期合作交流,学生将有机会前往世界一流的大学,如Monash大学、Monash大学苏州校区、墨尔本大学和京都大学等一流研究环境访问交流,从事前沿研究项目,获得有关技术的宝贵经验。
· 团队氛围融洽,学生将会和来自海内外的师兄师姐一起进步。团队成员来自于Monash大学,墨尔本大学,大连海事大学等国内外一流高校,你将获得优秀师兄师姐的帮助,助你冲击顶刊顶会。
· 团队将会为优秀毕业生进一步深造和发展提供强力的支持和推荐,优秀毕业生将有机会获得Monash大学(QS世界排名59)、墨尔本大学(QS世界排名37)等世界一流高校的全额奖学金,或者加盟燃石医学(上海)、百图生科等一流的互联网生物制药公司的机会。
欢迎满足下列条件的同学报考:
(1)具有较强的编程能力;
(2)对数据挖掘、机器学习和软件设计有浓厚兴趣。
(3)具有较好的英语水平;
此外,团队接受优秀的本科生参与科研训练,有意者请邮件或微信联系。
教育经历 · 2017年02月-2020年05月,澳大利亚蒙纳士大学,Biomedical Discovery Institute,生物信息学,博士
· 2013年09月-2016年06月,det365官方网站登录,det365官方网站登录,软件工程,工学硕士
· 2009年09月-2013年06月,det365官方网站登录,det365官方网站登录,软件工程,工学学士
工作经历 · 2022年06月-至今,det365官方网站登录,det365官方网站登录,教授
· 2020年09月-2022年06月,澳大利亚墨尔本大学,Peter Doherty感染与免疫研究所,生物信息学研究员
· 2020年05月-2020年09月,澳大利亚蒙纳士大学,生物医学发现研究所(BDI),博士后
· 2020年08月-2020年12月,澳大利亚蒙纳士大学,信息技术学院(FIT),数据科学助教
· 2018年10月-2020年05月,澳大利亚蒙纳士大学,生物医学发现研究所(BDI),生物信息学助研
· 2017年07月-2018年07月,澳大利亚蒙纳士大学,生物化学和分子生物学系,生物信息学助教
科研项目 · 国家自然科学基金青年项目,“蛋白酶特异性底物和裂解位点的生物信息学研究”,30万,主持
· 国家重点研发计划项目子课题,“基于人工智能的多组学数据分析平台构建”,50万,主持
· 陕西省秦创原引用高层次创新创业人才项目,“面向高靶点活性小分子发现的人工智能技术研究与平台搭建”,37万,主持
· det365官方网站登录人才科研经费,“数据驱动的生物大分子共价修饰生物信息学研究”,100万,主持
获奖情况 · 2023年,ISMB/ECCB 2023 Royal Society of Chemistry Best Poster Prize
· 2021年,FAOBMB青年科学家奖(Young Scientist Programme award)
· 2020年,Monash大学发表论文奖(Postgraduate Publications Award)
· 2020年,第十届生物科学,生物化学和生物信息学国际会议最佳报告奖(Best presentation award)
· 2020年,国家优秀自费留学生奖学金(全球500名)
· 2016年,Monash大学全额奖学金
学术兼职 · 担任国际期刊BMC Bioinformatics的编委;
· 担任国际期刊Frontiers in Bioinformatics的编辑;
· 担任国际期刊BioMed Research International的编辑;
· 担任国际期刊Current Gene Therapy的编辑;
· 担任国际期刊Artificial Intelligence and Applications的编辑
· 担任国际期刊International Journal of Molecular Sciences的客座编辑;
· 担任国际期刊Algorithms的客座编辑;
· 担任智能化农业装备学报青年编委;
· 曾担任第30届国际基因组信息学大会(GIW 2019)的程序委员会成员;
· 担任计算生物学和生物医学国际学术会议 (CBBS 2023)程序委员会委员
· 担任Bioinformatics, Briefings in Bioinformatics, BMC Bioinformatics, PLOS Computational Biology, IEEE/ACM Transactions on Computational Biology and Bioinformatics, Information Fusion和Pattern recognition等机器学习和生物信息学主流期刊的审稿人。
学术报告和国际会议 国际会议:
1. 第31届年度分子生物学智能系统和第22届欧洲计算生物学年会(The 31st Annual Intelligent Systems For Molecular Biology and the 22nd Annual European Conference on Computational Biology),2023年7月23-27日,大会报告:“ProsperousPlus: An integrated platform for protease-specific substrate cleavage prediction and machine learning model construction of more than 100 proteases”
2. 第二届电子信息工程与计算机技术国际会议 (The 2nd International Conference on Electronic Information Engineering and Computer Technology),陕西西安 (在线会议),2022年10月29日,主题报告 (Keynote Speaker): “Developing data-driven machine learning approaches to address bioinformatics classification problems”
3. 第16届亚洲及大洋洲生物化学与分子生物学家联盟会议 (FAOBMB 2021),新西兰基督城 (在线会议),2021年11月22-25日,分组报告
4. 第十届生物科学,生物化学和生物信息学国际会议 (ICBBB 2020),日本京都,2020年1月19-22日,特邀报告: “Leveraging the Power of Data-Driven Machine Learning Techniques to Address Significant Biomedical Classification Problems”.
5. 第十届生物科学,生物化学和生物信息学国际会议 (ICBBB 2020),日本京都,2020年1月19-22日,分组报告
6. 第一届国际人工智能会议 (A2IC 2018),西班牙巴塞罗那,2018年11月21-23日,分组报告
7. 第12届创新计算、信息和控制国际会议,日本久留米,2017年8月28-30日,分组报告
学术交流:
1. 2020年1月23日,日本京都大学生物信息学中心,学术报告:“A data-driven bioinformatic investigation into protein post-translational modifications”.
2. 2017年9月1日,日本京都大学生物信息学中心,学术报告:“PROSPERous: integrative high-throughput prediction of the substrate cleavage sites for 90 proteases with improved accuracy”.
学术成果 基因调控是现代分子生物学研究的中心课题之一。掌握基因调控机制对于了解动植物生长发育规律、形态结构特征及生物学功能至关重要。研究人员可根据生物分子在基因调控中的作用,描述人类疾病的诊断治疗的内在规律。迄今为止,科学界围绕中心法则这一主线相继发现和补充了多个新的调控途径和机制。在DNA层次上,有顺式作用元件、基因组调控序列和甲基化修饰等调控机制;在RNA层次上,有RNA编辑和转录后修饰等调控机制;在蛋白质层次上,有蛋白质翻译后的共价修饰等调控机制。本课题组研究主要聚焦在基因表达调控机制的生物信息学研究,具体的研究内容包括三方面:1. 蛋白质底物位点的共价修饰预测;2. DNA和RNA化学修饰位点识别;3. 基因组信号和调控元件注释。针对三个研究内容中存在的标签数据稀少和模型泛化能力弱等问题,进行建模和学习算法的创新,主要研究成果如图所示。
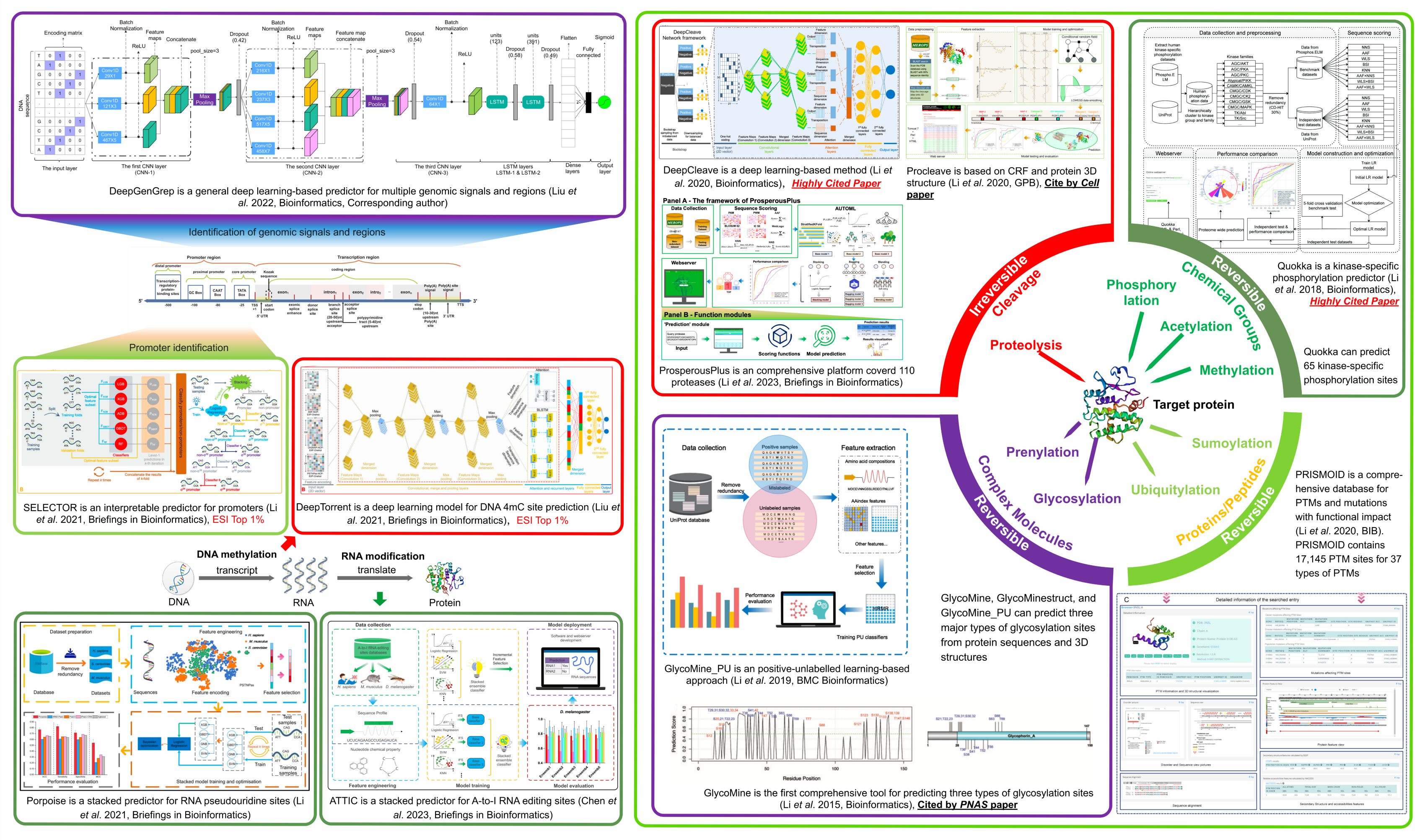
代表性学术论文如下 (*通讯作者,†并列一作):
1. Li F*, Wang C, Guo X, Akutsu T, Webb GI, Coin LJM, Kurgan L*, Song J*. “ProsperousPlus: An integrated platform for protease-specific substrate cleavage prediction and machine learning model construction of more than 100 proteases”. Briefings in Bioinformatics. 2023, 24(3), bbad170. (IF=13.994, 中科院大类一区, Top期刊, CCF B类, 西农G2期刊)
2. Li F, Chen J, Leier A, Marquez-Lago TT, Liu Q, Wang Y, Revote J, Smith AI, Akutsu T, Webb GI, Kurgan L, Song J. “DeepCleave: a deep learning predictor for caspase and matrix metalloprotease substrates and cleavage sites”. Bioinformatics. 2020, 36(4), 1057-1065. (IF=6.937, 中科院大类二区(小类一区), Top期刊, JCR Q1, CCF B类) (Clarivate高被引论文).
3. Li F, Li C, Marquez-Lago TT, Leier A, Akutsu T, Purcell AW, Smith AI, Lithgow T, Daly RJ, Song J. “Quokka: a comprehensive tool for rapid and accurate prediction of kinase family-specific phosphorylation sites in the human proteome”. Bioinformatics. 2018, 34(24), 4223-4231. (IF=6.937, 中科院大类二区(小类一区), Top期刊, JCR Q1, CCF B类)(Clarivate高被引论文).
4. Li F, Leier A, Liu Q, Wang Y, Xiang D, Akutsu T, Webb GI, Smith AI, Marquez-Lago TT, Li J, Song J. “Procleave: Predicting Protease-Specific Substrate Cleavage Sites by Combining Sequence and Structural Information”. Genomics, Proteomics & Bioinformatics. 2020, 18(1), 52-64. (IF=7.691, 中科院大类一区, Top期刊, JCR Q1).
5. Li F, Chen J, Ge Z, Wen Y, Yue Y, Hayashida M, Baggag A, Bensmail H, Song J. “Computational prediction and interpretation of both general and specific types of promoters in Escherichia coli by exploiting a stacked ensemble-learning framework”. Briefings in Bioinformatics. 2021, 22(2), 2126-2140. (IF=13.994, 中科院大类一区, Top期刊, JCR Q1, CCF B类) (Clarivate高被引论文).
6. Li F, Dong S, Leier A, Han M, Guo X, Xu J, Wang X, Pan S, Jia C, Zhang Y, Webb GI, Coin LJM, Li C, Song J. “Positive-unlabeled learning in bioinformatics and computational biology: a brief review”. Briefings in Bioinformatics. 2021, 23(1), bbab461. (IF=13.994, 中科院大类一区, Top期刊, JCR Q1, CCF B类).
7. Li F, Guo X, Jin P, Chen J, Xiang D, Song J, Coin LJM. “Porpoise: a new approach for accurate prediction of RNA pseudouridine sites”. Briefings in Bioinformatics. 2021, 22(6), bbab245. (IF=13.994, 中科院大类一区, Top期刊, JCR Q1, CCF B类).
8. Li F, Fan C, Marquez-Lago TT, Leier A, Revote J, Jia C, Zhu Y, Smith AI, Webb GI, Liu Q, Wei L, Li J, Song J. “PRISMOID: a comprehensive 3D structure database for post-translational modifications and mutations with functional impact”. Briefings in Bioinformatics. 2020, 21(3), 1069-1079. (IF=13.994, 中科院大类一区, Top期刊, JCR Q1, CCF B类).
9. Li F, Wang Y, Li C, Marquez-Lago TT, Leier A, Rawlings ND, Haffari G, Revote J, Akutsu T, Chou KC, Purcell AW, Pike RN, Webb GI, Smith AI, Lithgow T, Daly RJ, Whisstock JC, Song J. “Twenty years of bioinformatics research for protease-specific substrate and cleavage site prediction: a comprehensive revisit and benchmarking of existing methods”. Briefings in Bioinformatics. 2019, 20(6), 2150-2166. (IF=11.622, 中科院大类一区, Top期刊, JCR Q1, CCF B类).
10. Li F, Li C, Wang M, Webb GI, Zhang Y, Whisstock JC, Song J. “GlycoMine: a machine learning-based approach for predicting N-, C-, and O-linked glycosylation in the human proteome”. Bioinformatics. 2015, 31(9), 1411-1419. (IF=6.937, 中科院大类二区(小类一区), Top期刊, JCR Q1, CCF B类).
11. Li F, Zhang Y, Purcell AW, Webb GI, Chou KC, Lithgow T, Li C, Song J. “Positive-unlabelled learning of glycosylation sites in the human proteome”. BMC Bioinformatics. 2019, 20, 112. (IF=3.169, 中科院大类三区(小类二区), JCR Q1, CCF C类).
12. Li F, Guo X, Xiang D, Pitt EM, Bainomugisa A, Coin LJM. “Computational analysis and prediction of PE_PGRS proteins using machine learning”. Computational and Structural Biotechnology Journal. 2022. 20, 662-674. (IF=7.271, 中科院大类二区, JCR Q1).
13. Li F*, Guo X, Bi Y, Jia R, Pitt ME, Pan S, Li S, Gasser RB, Coin LJM*, Song J*. “Digerati – a multipath parallel hybrid deep learning framework for the identification of mycobacterial PE/PPE proteins”. Computers in Biology and Medicine. 2023, 163, 107155. (IF=6.698, 中科院大类二区, 小类一区)
14. Liu Q, Chen J, Wang Y, Li S, Jia C*, Song J*, Li F*. “DeepTorrent: a deep learning-based approach for predicting DNA N4-methylcytosine sites”. Briefings in Bioinformatics. 2021, 22(3), bbaa124. (IF=13.994, 中科院大类一区, Top期刊, CCF B类) (Clarivate高被引论文).
15. Wang C, He Z, Jia R, Pan S, Coin LJM, Song J, Li F*. “PLANNER: a multi-scale deep language model for the origins of replication site prediction”. IEEE Journal of Biomedical and Health Informatics. 2024, doi: 10.1109/JBHI.2024.3349584. (IF=7.7, 中科院大类一区, Top期刊)
16. Chen R†, Li F*†, Guo X, Bi Y, Li C, Pan S, Coin LJM*, Song J*. “ATTIC is an integrated approach for predicting A-to-I RNA editing sites in three species”. Briefings in Bioinformatics. 2023, 24(3), bbad170. (IF=13.994, 中科院大类一区, Top期刊, CCF B类)
17. Zhu Y†, Li F*†, Guo X, Wang X, Coin LJM, Webb GI, Song J*, Jia C*. “TIMER is a Siamese neural network-based framework for identifying both general and species-specific bacterial promoters”. Briefings in Bioinformatics. 2023, in press. (IF=13.994, 中科院大类一区, Top期刊, CCF B类)
18. Liu Q, Fang H, Wang M, Li S, Coin LJM, Li F*, Song J*. “DeepGenGrep: a general deep learning-based predictor for multiple genomic signals and regions”. Bioinformatics. 2022, 38(17):4053–4061. (IF=6.937, 中科院大类二区(小类一区), Top期刊, JCR Q1, CCF B类)
19. Bi Y, Li F*, Guo X, Wang Z, Pan T, Guo Y, Webb GI, Yao J, Jia C*, Song J*. “Clarion is a multi-label problem transformation method for identifying mRNA subcellular localisations”. Briefings in Bioinformatics. 2022, in press. (IF=13.994, 中科院大类一区, Top期刊, CCF B类)
20. Peng X, Wang X, Guo Y, Ge Z*, Li F*, Gao X*, Song J*. “RBP-TSTL is a two-stage transfer learning framework for genome-scale prediction of RNA-binding proteins”. Briefings in Bioinformatics. 2022, bbac215. (IF=13.994, 中科院大类一区, Top期刊, CCF B类).
21. Jia C, Bi Y, Chen J, Leier A, Li F*, Song J*. “PASSION: an ensemble neural network approach for identifying the binding sites of RBPs on circRNAs”. Bioinformatics. 2020, 36(15), 4276-4282. (IF=6.937, 中科院二区(小类一区), Top期刊, JCR Q1, CCF B类).
22. Wang X, Li F*, Xu J, Rong J, Webb GI, Ge Z*, Li J, Song J*. “ASPIRER: a new computational approach for identifying non-classical secreted proteins based on deep learning”. Briefings in Bioinformatics. 2022, 23(2), bbac031.(IF=13.994, 中科院大类一区, Top期刊, CCF B类)
23. Jia C, Zhang M, Fan C, Li F*, Song J*. “Formator: predicting lysine formylation sites based on the most distant undersampling and safe-level synthetic minority oversampling”. IEEE/ACM transactions on computational biology and bioinformatics. 2021 18(5), 1937-1945. (IF=3.71, 中科院三区(小类二区), JCR Q1, CCF B类).
24. Chai D, Jia C*, Zheng J, Zou Q, Li F*. “Staem5: a novel computational approach for accurate prediction of m5C site”. Molecular Therapy-Nucleic Acids. 2021, 26, 1027-1034. (IF=8.886, 中科院大类二区, JCR Q1).
25. Bu Y, Jia C, Guo X, Li F*, Song J*. “COPPER: an ensemble deep-learning approach for identifying exclusive virus-derived small interfering RNAs in plants”. Briefings in Functional Genomics. 2022, elac049. (IF=4.840, JCR Q1).
26. Bi Y, Xiang D, Ge Z, Li F*, Jia C*, Song J*. “An interpretable prediction model for identifying N7-methylguanosine sites based on XGBoost and SHAP”. Molecular Therapy-Nucleic Acids. 2020, 22, 362-372. (IF=8.886, 中科院大类二区, JCR Q1).
27. Song J†, Li F†, Leier A, Marquez-Lago TT, Akutsu T, Haffari G, Chou KC, Webb GI, Pike RN. “PROSPERous: high-throughput prediction of substrate cleavage sites for 90 proteases with improved accuracy”. Bioinformatics. 2017, 34(4), 684-687. (IF=6.937, 中科院大类二区(小类一区), Top期刊, JCR Q1, CCF B类) (Clarivate高被引论文).
28. Mei S†, Li F†, Xiang D, Ayala R, Faridi P, Webb GI, Illing PT, Rossjohn J, Akutsu T, Croft NP, Purcell AW, Song J. “Anthem: a user customised tool for fast and accurate prediction of binding between peptides and HLA class I molecules”. Briefings in Bioinformatics. 2021, 22(5), bbaa415. (IF=13.994, 中科院大类一区, Top期刊, JCR Q1, CCF B类).
29. Liang X†, Li F†, Chen J, Li J, Wu H, Li S, Song J, Liu Q. “Large-scale comparative review and assessment of computational methods for anti-cancer peptide identification”. Briefings in Bioinformatics. 2021, 22(4), bbaa312. (IF=13.994, 中科院大类一区, Top期刊, JCR Q1, CCF B类).
30. Yan Zhu†, Li F†, Xiang D, Akutsu T, Song J, Jia C. “Computational identification of eukaryotic promoters based on cascaded deep capsule neural networks”. Briefings in Bioinformatics. 2021, 22(4), bbaa299. (IF=13.994, 中科院大类一区, Top期刊, JCR Q1, CCF B类).
31. Jia X†, Zhao P†, Li F†, Qin Z, Ren H, Li J, Miao C, Zhao Q, Akutsu T, Dou G, Chen Z, Song J. “ResNetKhib: a novel cell type-specific tool for predicting lysine 2-hydroxyisobutylation sites via transfer learning”. Briefings in Bioinformatics. 2023, 24(2), bbad063. (IF=13.994, 中科院大类一区, Top期刊, CCF B类)
32. Zhang M†, Jia C†, Li F†, Li C†, Zhu Y, Akutsu T, Webb GI, Zou Q, Coin JML, Song J. “Critical assessment of computational tools for prokaryotic and eukaryotic promoter prediction”. Briefings in Bioinformatics. 2022, bbab551. (IF=13.994, 中科院大类一区, Top期刊, JCR Q1, CCF B类).
33. Zhang M†, Li F†, Marquez-Lago TT, Leier A, Fan C, Chou KC, Song J, Jia C. “MULTiPly: a novel multi-layer predictor for discovering general and specific types of promoters”. Bioinformatics. 2019, 35(17), 2957-2965. (IF=6.937, 中科院大类二区, Top期刊, JCR Q1, CCF B类) (Clarivate高被引论文).
34. Ma X, Zhang L, Song J, Nguyen E, Lee SR, Rodgers SJ, Li F, Huang C, Schittenhelm RB, Chan H, Chheang C, Wu J, Brown KK, Mitchell CA, Simpson KJ, Daly RJ. “Characterization of the Src-regulated kinome identifies SGK1 as a key mediator of Src-induced transformation”. Nature Communications. 2019, 10, 296. (IF=14.919, 中科院大类一区, JCR Q1).
课题组成员 博士生:
郭旭东,王聪
硕士生:
贾润昌,冉子旭,郝亿,贾盼
已毕业硕士生:
Ruyi Chen (墨尔本大学生物信息学专业硕士毕业,现于昆士兰大学全额奖学金攻读博士学位)
张萌(协助大连海事大学贾藏芝教授指导,现于南京航空航天大学攻读博士学位)
朱燕(协助大连海事大学贾藏芝教授指导,现于哈尔滨工业大学攻读博士学位)
Xiaolan Tan(与澳大利亚格里菲斯大学潘世瑞教授共同指导,现于Monash大学攻读博士学位)
向东旭(2020届det365官方网站登录毕业生,邀请赴Monash访问学习一年,现于墨尔本大学攻读硕士学位)